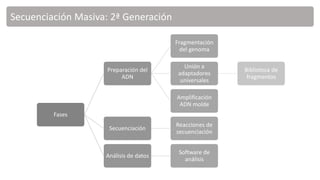
El documento describe los métodos manuales de secuenciación de ácidos nucleicos, incluyendo el método químico de Maxam y Gilbert y el método enzimático de Sanger y Coulson. El método de Maxam y Gilbert usa sustancias químicas para fragmentar el ADN, mientras que el método de Sanger y Coulson usa la ADN polimerasa. Ambos métodos generan fragmentos de diferentes tamaños que pueden separarse y leerse para determinar la secuencia de nucleótidos. El documento también explica los principios y procedim