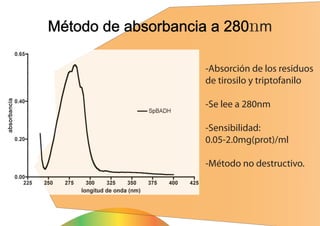
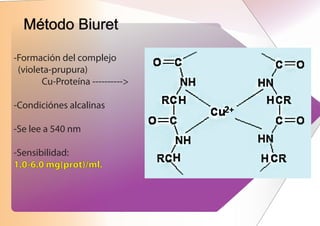
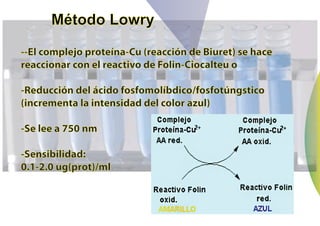
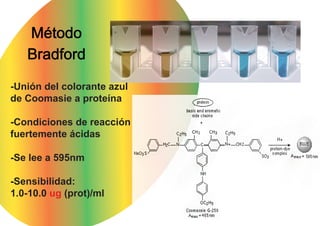
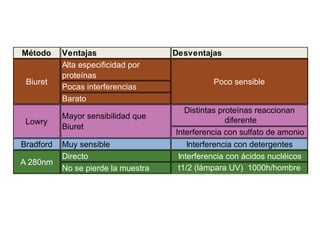
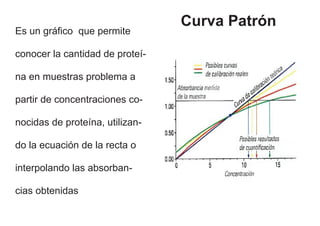
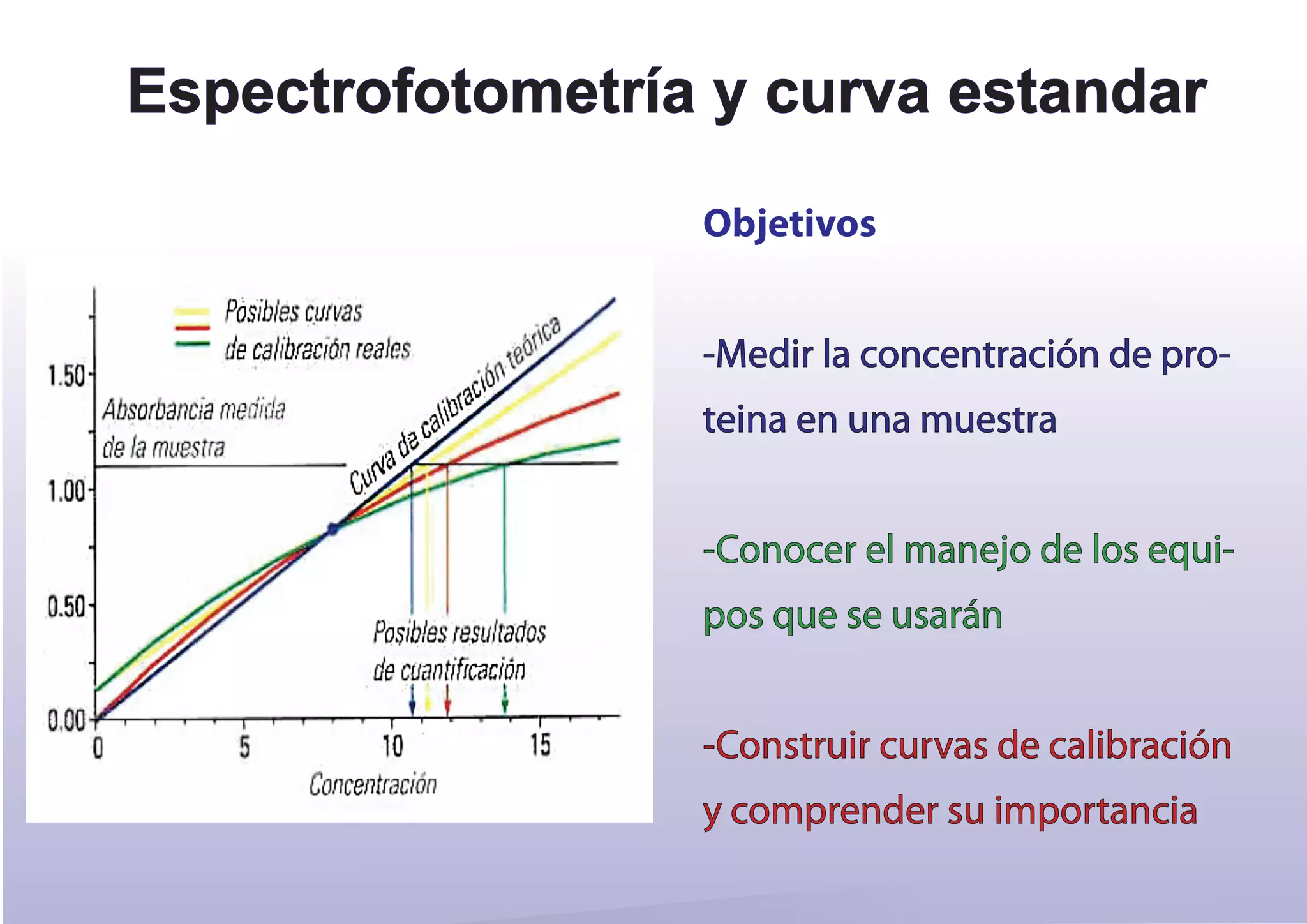
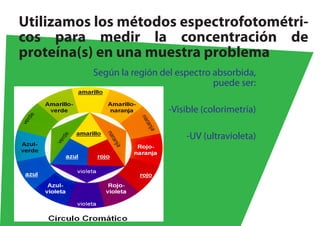
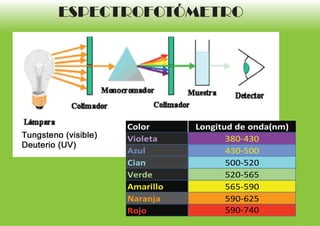
El documento describe métodos para medir la concentración de proteínas, incluyendo espectrofotometría y curvas de calibración. Explica cómo construir una curva estándar midiendo la absorbancia de muestras con concentraciones conocidas de proteína y usarla para determinar la concentración de proteínas en muestras problema. También compara métodos como Biuret, Lowry, Bradford y absorbancia a 280 nm, discutiendo sus ventajas y desventajas.
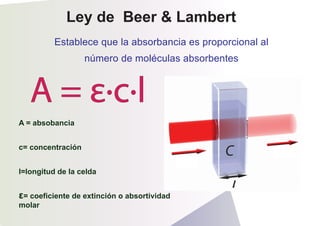
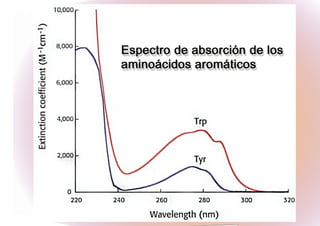
![Coeficiente de extinción
o absortividad molar
Es una propiedad intrínseca de los compuestos. Es una medida de
absorción de las sustancias a una determinada longitud de onda
Unidades: ε[=]M-1cm-1
F o Phe 260nm W o Trp 295 nm
Y o Tir 275nm](https://image.slidesharecdn.com/espectro-110214003027-phpapp01/85/Espectro-5-320.jpg)