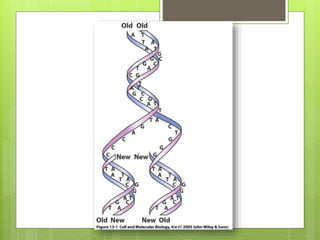
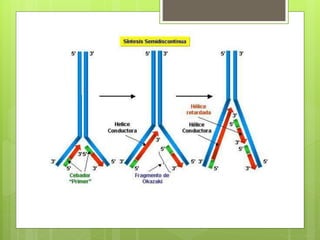
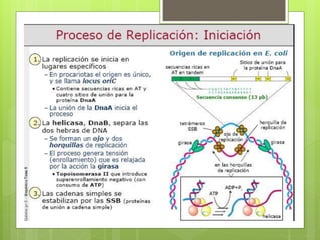
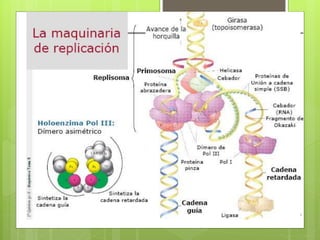
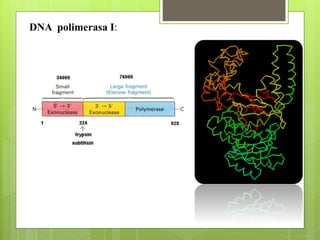
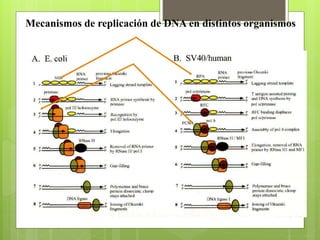
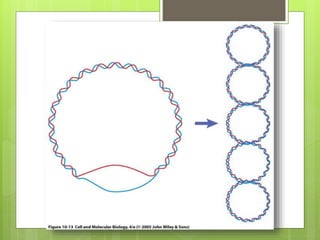
El documento describe la estructura y replicación del cromosoma bacteriano. El cromosoma bacteriano generalmente consiste en un solo cromosoma circular compuesto de ADN, ARN y proteínas. La replicación del cromosoma ocurre de forma bidireccional a partir de un origen de replicación y requiere enzimas como la ADN helicasa, ADN polimerasas y proteínas estructurales como HU.























![En una molécula de B-DNA relajada L= T= (nº pb)/10,4.
Una hebra gira sobre la otra cada 10,4 pares de bases y
un polinucleótido da una vuelta completa en torno al
eje cada 10,4 pb.
Si el valor de 10,4 pares de bases por vuelta se altera, la
molécula de B-DNA se encuentra sometida a una
tensión. Aún así, la estructura se puede forzar para que
T sea igual a [(nº pb)/10,4] mediante una variación de
W: La doble hélice se “retuerce” (superenrollamiento).
En este caso T será distinto de L. (T=L-W)
Procesos que conducen a que T sea distinto de [(nº
pb)/10,4].
En función de la secuencia de nucleótidos se puede
optar por otras alternativas al superenrollamiento para
forzar que T sea igual a [(nº pb)/10,4]:](https://image.slidesharecdn.com/nucleoi-140619152020-phpapp02/85/Nucleoi-25-320.jpg)