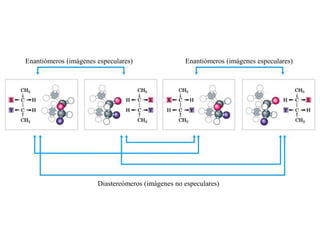
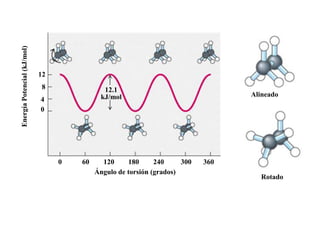
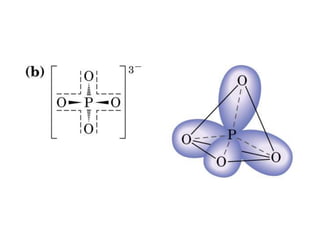
El documento presenta una tabla con diferentes grupos funcionales encontrados en biomoléculas, incluyendo alcoholes, aldehídos, aminas, ésteres, tioésteres y ácidos carboxílicos. También incluye información sobre los números de electrones desapareados y los electrones de la última capa de varias moléculas como el agua, amoníaco y metano.