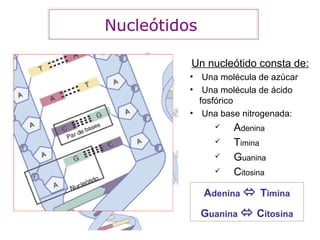
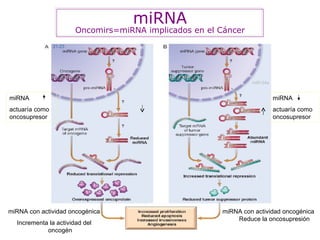
1) El documento describe las bases moleculares del cáncer y la conversión de protooncogenes en oncogenes. 2) Un protooncogen es un gen normal que regula el crecimiento celular, mientras que un oncogén es una versión mutada del protooncogen que causa proliferación celular descontrolada. 3) Los oncogenes se activan por mutaciones en el ADN causadas por factores como radiación, químicos o virus, o por alteraciones epigenéticas como la metilación del ADN.