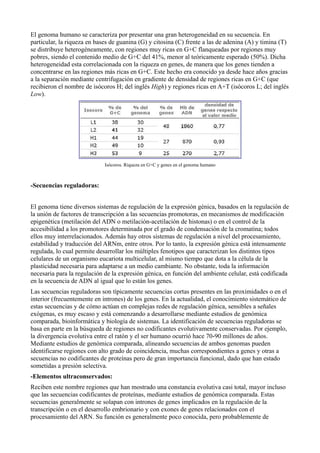
El documento resume la historia de la genética y el descubrimiento del genoma humano. Comenzó con el trabajo de Gregor Mendel en 1866 sobre la herencia de rasgos en plantas, que estableció las bases de la genética mendeliana. En 1953, Watson y Crick determinaron que el ADN tiene una estructura de doble hélice, lo que marcó el comienzo de la era de la genética molecular. El Proyecto Genoma Humano secuenció completamente el genoma humano para 2003, identificando entre 20,000-25,000 genes en los 23