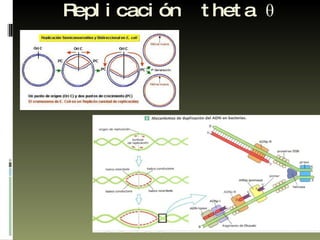
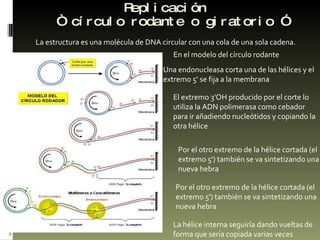
Este documento describe la replicación del ADN en procariotas. 1) El ADN procariótico es circular y sin nucleosomas. 2) La replicación es bidireccional desde un único origen de replicación y mantiene la semiconservación de las hebras. 3) La replicación implica las fases de iniciación, elongación y terminación catalizadas por distintas proteínas como la ADN polimerasa y la helicasa.