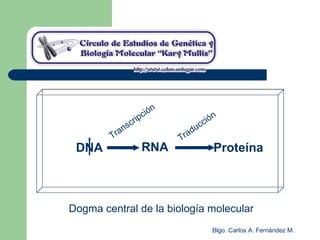
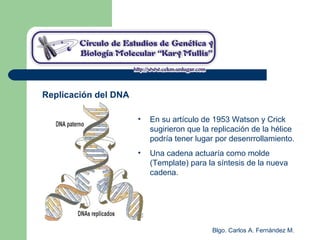
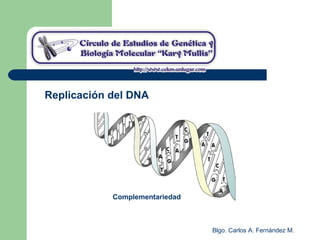
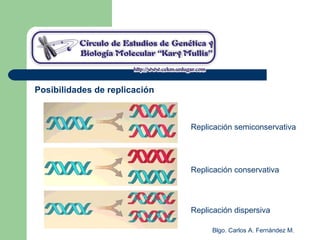
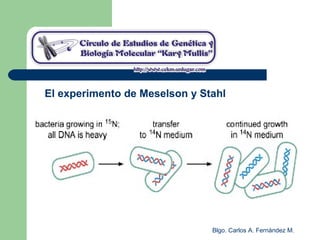
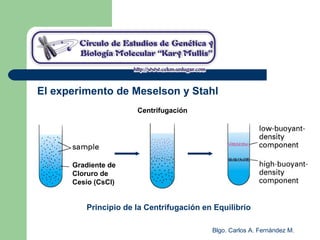
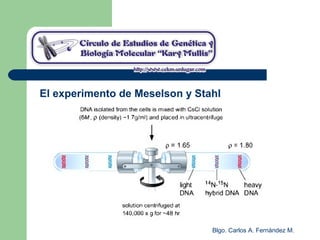
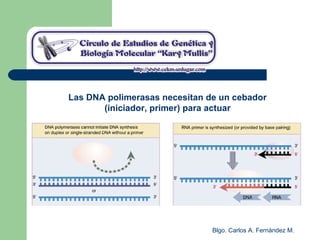
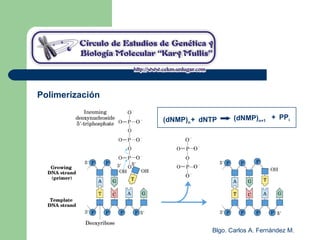
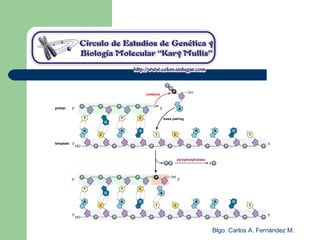
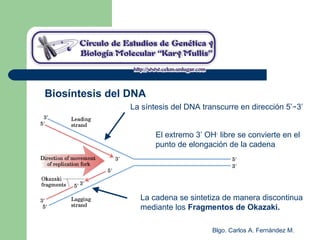
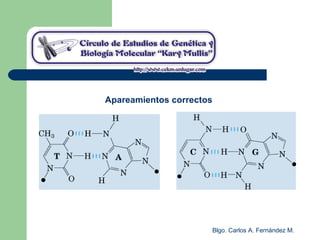
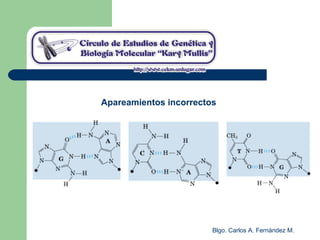
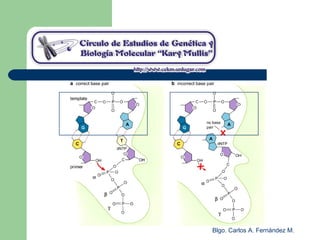
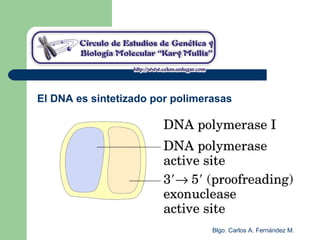
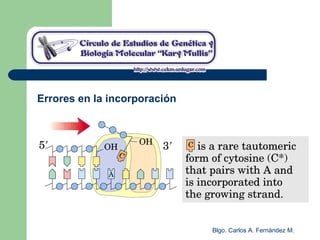
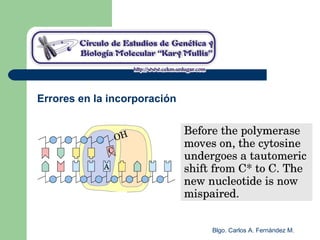
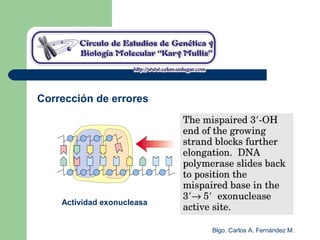
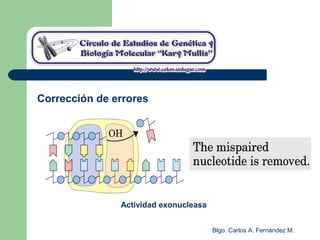
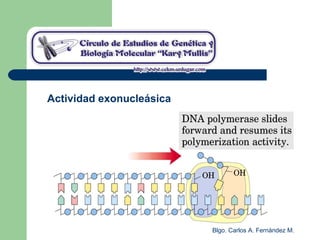
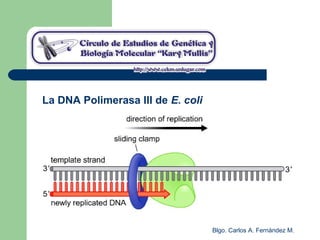
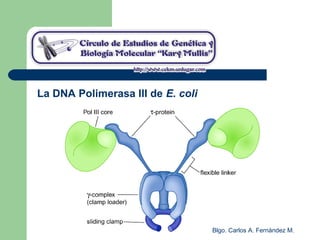
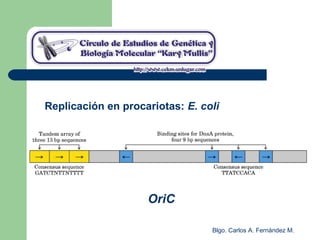
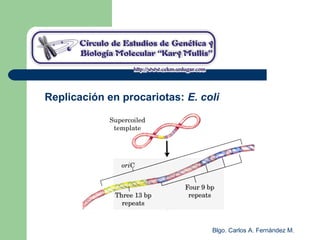
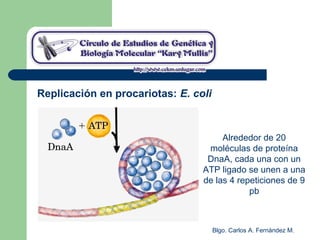
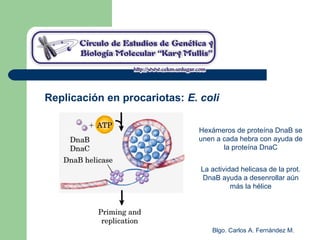
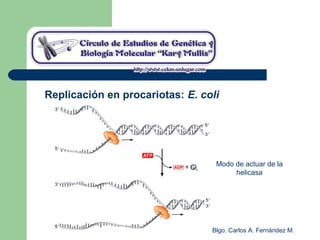
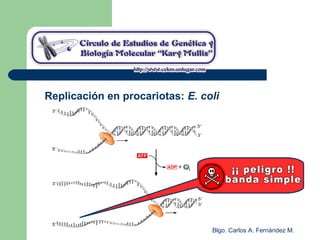
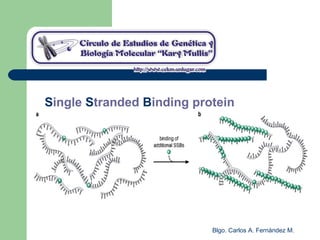
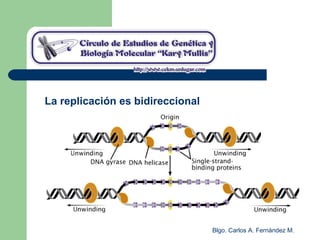
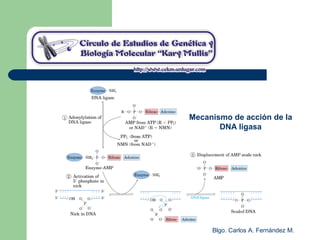
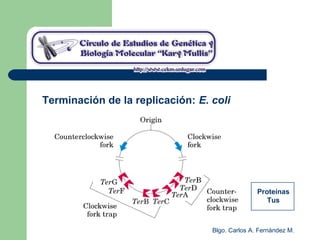
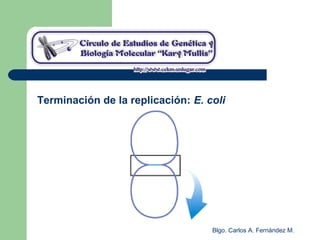
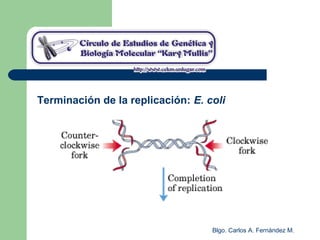
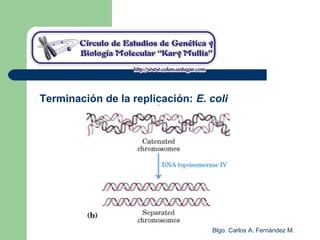
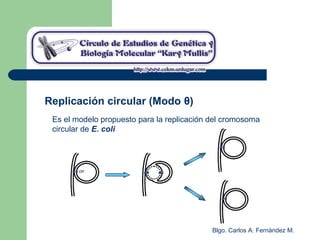
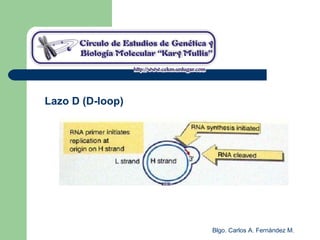
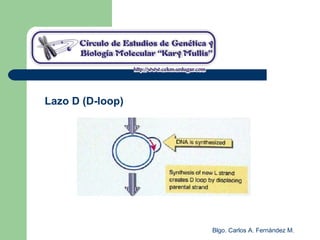
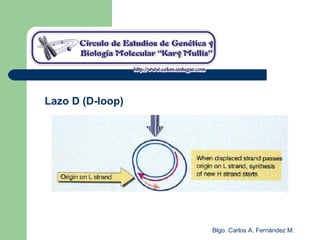
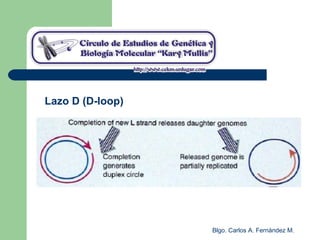
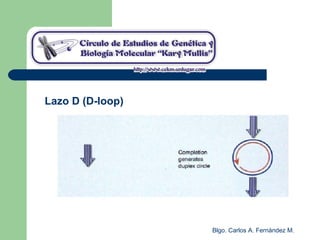
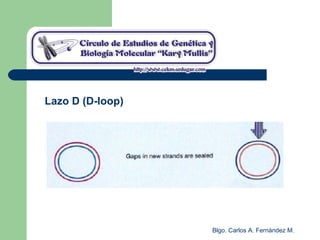
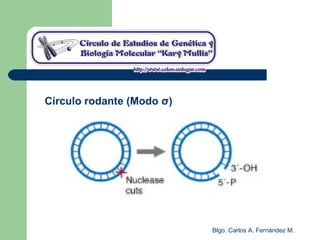
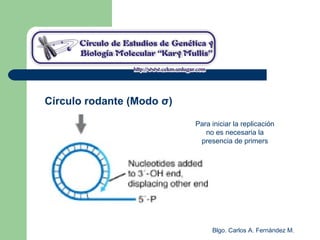
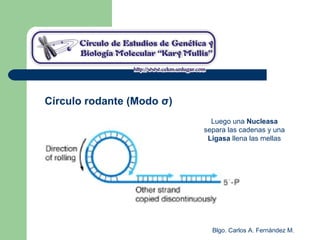
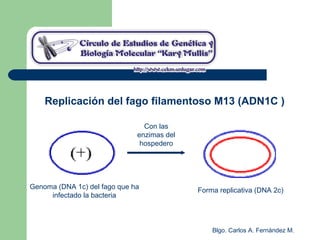
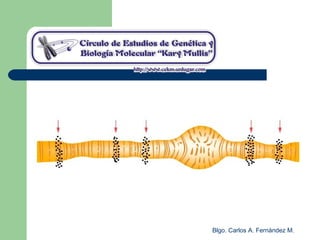
El documento describe los mecanismos de replicación del ADN en procariotas. Explica que el ADN es sintetizado por polimerasas siguiendo las reglas de complementariedad de Watson y Crick, y que la replicación en E. coli ocurre de forma bidireccional desde un origen de replicación y utiliza proteínas como la helicasa DnaB y la proteína de unión a ADN simple cadena. También resume los tres modelos de replicación circular en procariotas: modo θ en E. coli, lazo D en mitocondrias y cloroplastos, y