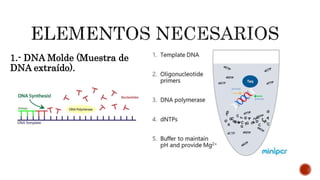
La reacción en cadena de la polimerasa (PCR) es una técnica que permite amplificar segmentos pequeños de ADN. Involucra la desnaturalización del ADN, la hibridación de cebadores y la extensión mediante una ADN polimerasa, lo que genera millones de copias del fragmento de interés en unas pocas horas. Se utiliza ampliamente en investigación, diagnóstico médico y aplicaciones forenses.