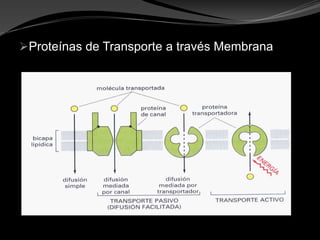
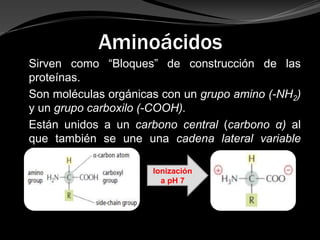
El documento describe las proteínas, incluyendo que son macromoléculas compuestas por cadenas polipeptídicas plegadas en una forma tridimensional característica. Discuten las diferentes funciones, estructuras y tipos de proteínas, así como los aminoácidos que son los bloques de construcción de las proteínas. También cubren los procesos de plegamiento de proteínas y desnaturalización.