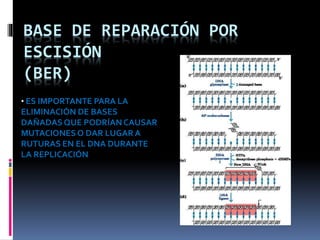
La función de la reparación del ADN es mantener la información genética intacta. Existen mecanismos como la reparación por escisión de bases y la recombinación homóloga que reparan los daños al ADN causados por factores ambientales y procesos metabólicos, los cuales ocurren a una tasa de entre 1,000 y 1 millón de lesiones por célula por día. Las lesiones no reparadas pueden causar mutaciones e impedir la función celular, aumentando el riesgo de cáncer.