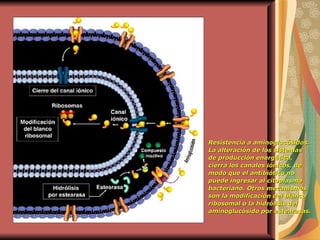
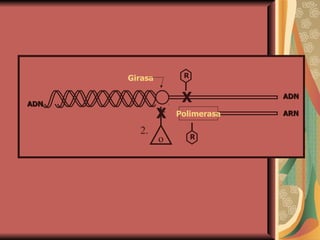
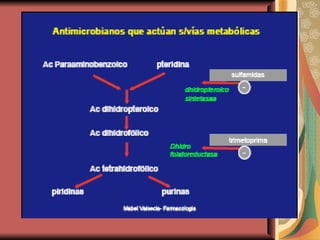
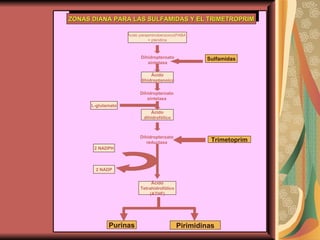
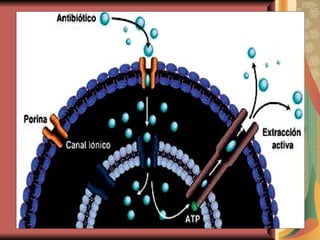
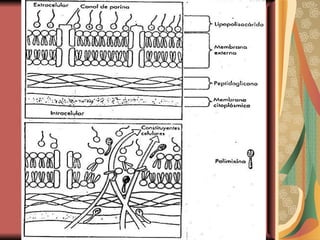
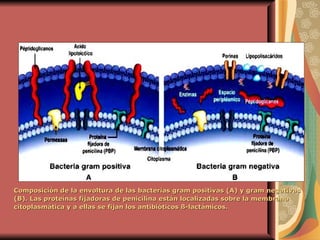
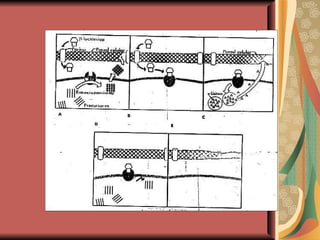
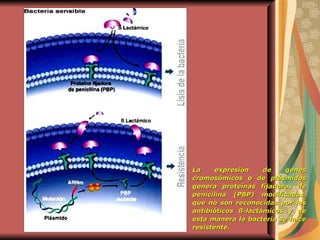
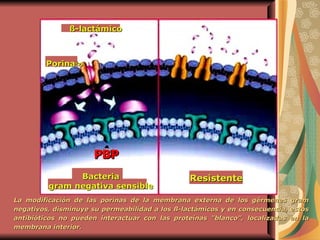
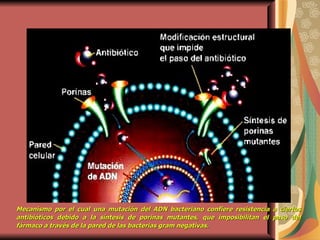
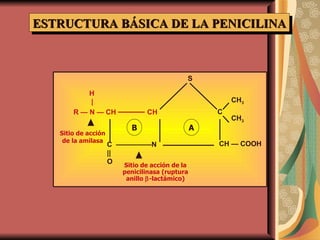
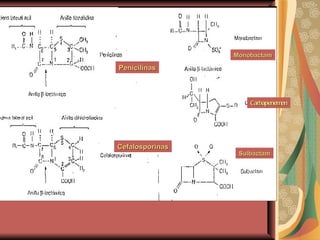
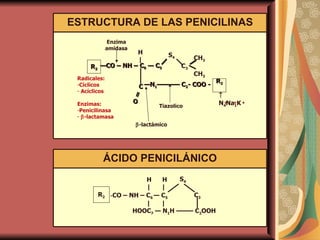
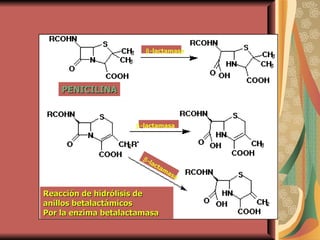
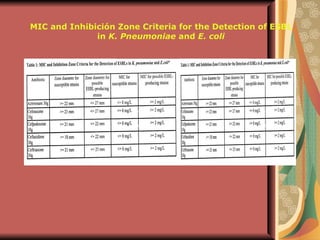
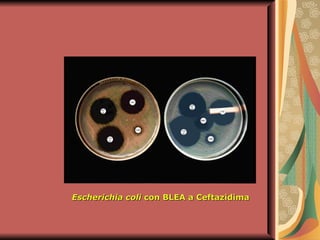
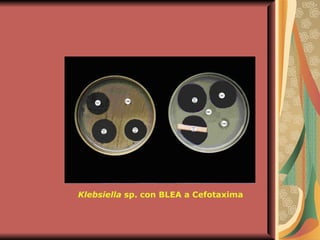
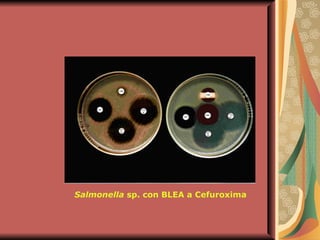
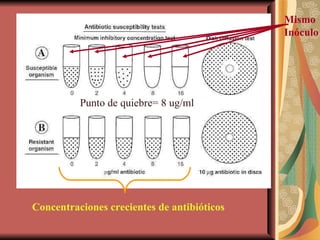
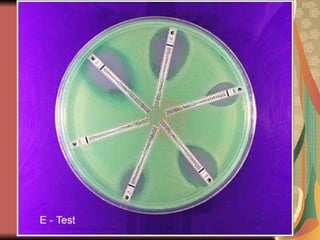
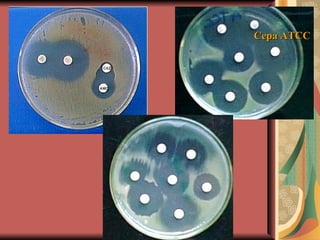
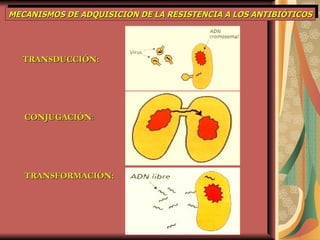
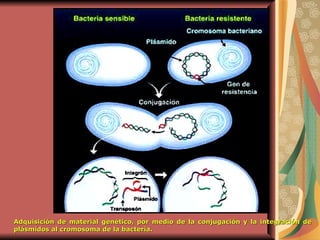
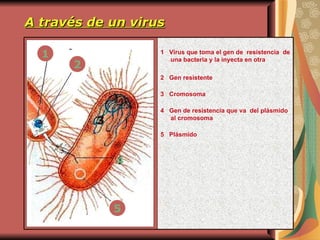
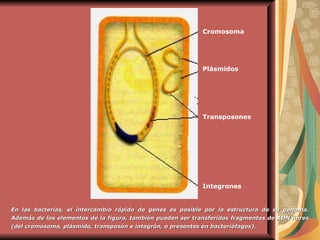
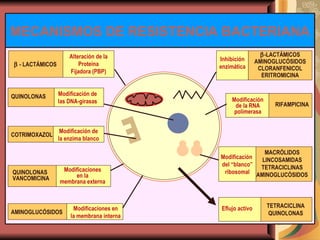
El documento describe los mecanismos por los cuales las bacterias adquieren resistencia a los antibióticos, incluyendo la transferencia de genes a través de conjugación, transformación y transducción. También explica los diferentes mecanismos de resistencia como la modificación de proteínas diana, bombas de eflujo activo, y la producción de enzimas que inactivan antibióticos. Finalmente, proporciona detalles sobre técnicas para detectar betalactamasas y realizar pruebas de sensibilidad antibiótica.






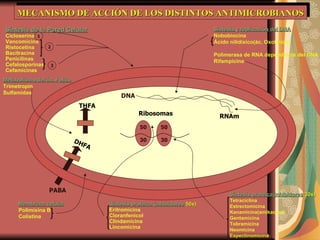

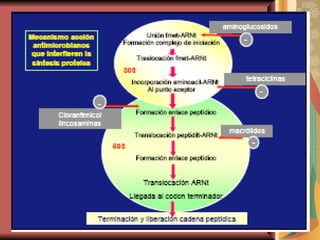
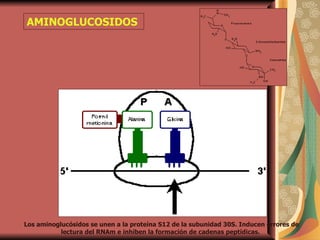
![Unión del fmet ARNt y formación del complejo de iniciación en el ribosoma 70S Translocación del fmet ARNt desde el punto aceptor [A] hasta el punto donante [P] Unión de nuevo aminoacil ARNt (1) al punto aceptor Formación de enlace peptídico catalizada por peptidil transferasa Liberación de ARNt no cargado AMINOGLUCÓSIDOS TETRACICLINAS CLORANFENICOL LINCOSAMIDAS ERITROMICINA Translocación del peptidil ARNt (1) Unión de aminoacil ARNt (2) Formación de enlace peptídico, elongación de cadena peptídica Transformación del peptidil ARNt expone el codón terminador Terminación y liberación de la cadena peptídica ÁCIDO FUSÍDICO INHIBIDORES DE LA SÍNTESIS DE PROTEINAS Reacciones diana inhibidas Agentes antimicrobianos](https://image.slidesharecdn.com/resistenciabacteriana-110306204539-phpapp02/85/Resistencia-bacteriana-15-320.jpg)