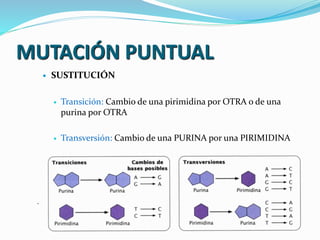
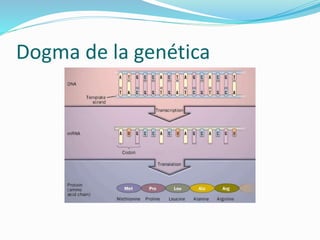
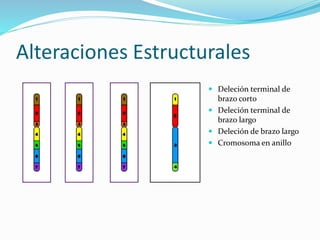
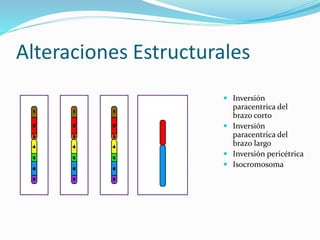
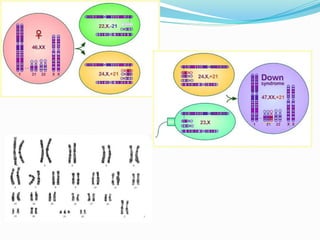
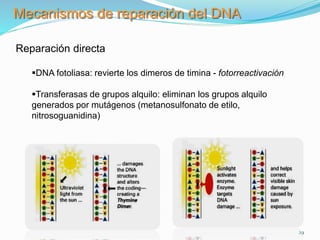
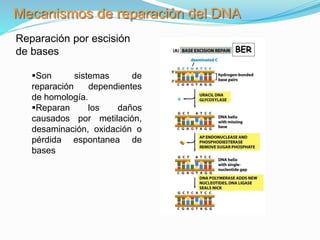
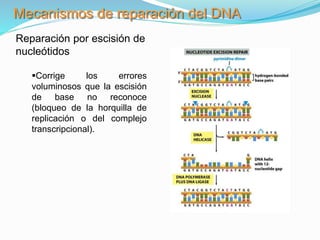
La mutación se define como un cambio permanente en la secuencia de bases de un organismo. Puede ser molecular, afectando el ADN de forma puntual, o cromosómica, mediante alteraciones estructurales o del número de cromosomas. Las mutaciones moleculares incluyen sustituciones, deleciones e inserciones de bases y pueden ser silenciosas, missense o nonsense dependiendo de cómo afecten a la proteína resultante.