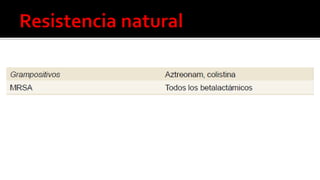
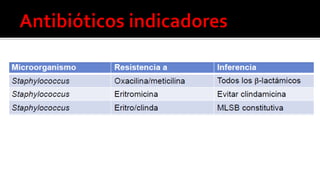
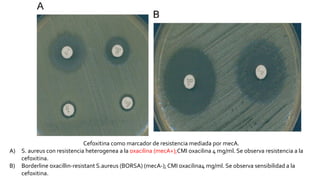
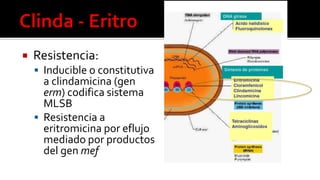
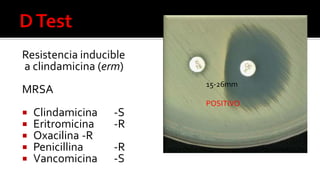
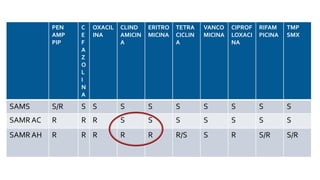
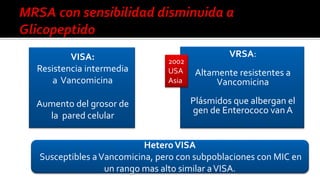
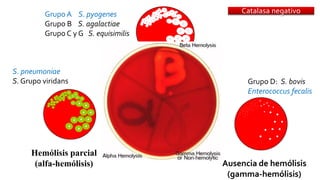
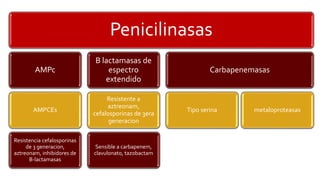
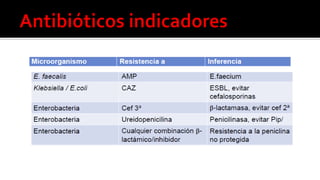
Este documento describe diferentes mecanismos de resistencia a antibióticos β-lactámicos en bacterias Gram-positivas y Gram-negativas, incluyendo la producción de betalactamasas, la sobreexpresión de proteínas de unión a penicilina, y la adquisición de genes de resistencia. Explica cómo estos mecanismos conducen a perfiles de resistencia específicos y ofrece recomendaciones de tratamiento basadas en los patrones de susceptibilidad.